
Hai anak teknik, khususnya untuk kamu anak teknik kimia, lingkungan, ataupun pangan, pastinya satu diantara kalian pernah mendengar penelitian menarik terkait dengan mikroba kan. Dimulai dari mikroba yang mampu mengurai plastik, mikroba yang mampu mengurai minyak, ataupun mikroba yang mampu mengolah lahan bekas tambang. Saya yakin ada banyak anak teknik yang mengagumi penemuan-penemuan tersebut, termasuk saya hehe.
Penelitian-penelitian tersebut memang merupakan trend penelitian yang saat ini berkembang di dunia bioteknologi. Namun, tidak menutup kemungkinan bahwa anak teknik juga bisa melakukannya lho. Sebagai anak teknik, kita bisa memanfaatkan penelitian tersebut untuk mengembangkan proses atau teknik berbasis biologi, yang pastinya akan bermanfaat untuk berbagai bidang, seperti energi, ekonomi, pangan, ataupun lingkungan. Tapi kendala yang pastinya kita hadapi adalah berkaitan dengan bagaimana cara mendapatkan informasi terkait fungsi tertentu mikroba, tanpa harus melakukan eksperimen kembali yang tentu memakan banyak waktu dan uang.
Saat ini, telah tersedia database-database yang menyediakan informasi terkait jalur metabolisme mikroba untuk memahami bagaimana mikroba-mikroba mengkonversi suatu bahan menjadi bahan lainnya, salah satunya dikenal sebagai RAST. Apa itu RAST? bagaimana cara kerjanya? Dan bagaimana cara menggunakannya? Semua informasi tersebut akan dijelaskan secara lengkap dalam artikel ini, selamat membaca :).
Mengenal RAST (Rapid Annotation using Subsystem Technology)
RAST, atau Rapid Annotation using Subsystem Technology, adalah alat komputasi yang dirancang untuk anotasi cepat genom mikroba. RAST umumnya menyediakan anotasi otomatis berkualitas tinggi terhadap fitur genom, yang memungkinkan peneliti memahami peran fungsional gen dan potensi metabolik suatu organisme.
Bagi anda yang masih asing dengan istilah-istilah tersebut, berikut saya jelaskan secara singkat terkait genom dan anotasi. Genom adalah istilah yang sering digunakan untuk mewakili satu paket lengkap materi genetik (berupa DNA ataupun RNA). Pada umumnya, genom mengandung semua instruksi yang dibutuhkan oleh organisme untuk tumbuh, berkembang, berinteraksi, dan bereproduksi. Genom memiliki beberapa komponen utama yaitu gen, non-coding DNA, senyawa regulatory, dan sekuen-sekuen repetitif. Gen merupakan bagian dari DNA yang mengkode protein atau RNA fungsional. Non-coding DNA merupakan bagian DNA yang tidak mengkode protein, tapi bermanfaat untuk regulasi ekspresi gen. Contoh non-coding DNA adalah enhancers, promoters, dan introns. Sedangkan senyawa Regulatory berfungsi mengontrol ekspresi gen pada suatu sistem.
Bagaimana dengan anotasi? Anotasi, dalam bioinformatika, mengacu kepada identifikasi dan deskripsi dari senyawa fungsional pada sekuens biologi, seperti DNA,RNA, atau Protein. Apa sih sekuens itu? Sekuens itu adalah urutan linear dari nukleotida dalam DNA/RNA (yang biasanya dikenal sebagai ATCG) atau asam amino dalam protein. Pada dasarnya sekuens mengandung informasi biologis yang menentukan fungsi genetik dan struktur protein. Anotasi pada umumnya terdiri atas dua jenis, yaitu anotasi struktural dan anotasi fungsional. Anotasi struktural mengandung fitur genomik seperti gen-gen, intron, exon, senyawa regulatory, dan sekuens repetitif. Sedangkan anotasi fungsional umumnya mengandung peran biologis dari gen dan protein, seperti fungsi protein dan jalurnya, peran metabolik dan klasifikasi enzim, serta interaksi antar protein.
Bagaimana sudah paham terkait gen dan anotasi? Jika sudah paham, yuk kembali ke pembahasan tentang RAST. Pada dasarnya, ada beberapa fitur utama pada RAST dalam memproses dan menyajikan informasi terkait anotasi genom yang kita inginkan. Fitur yang pertama adalah RAST mampu melakukan anotasi genom secara otomatis, sehingga RAST dapat mengidentifikasi gen pengkode protein, gen RNA, dan fitur lainnya, serta dapat memberikan peran fungsional berdasarkan subsistem yang dikurasi. Selain itu, anotasi pada RAST berbasis subsistem, yang memungkinkan RAST mengelompokkan gen ke dalam subsistem fungsional, seperti metabolisme, transportasi, dan regulasi. Anotasi ini juga memungkinkan RAST untuk melakukan analisis tingkat jalur metabolisme. RAST juga memiliki fungsi yang memungkinkan perbandingan antar genom yang telah dianotasi untuk mengidentifikasi fitur unik dan yang sama, memiliki interface yang interaktif, dan terintegrasi dengan database SEED, database bioinformatika untuk anotasi genom dan komparasi genomik.
Tutorial Penggunaan RAST
Pada bagian ini, akan dijelaskan terkait cara menggunakan RAST berdasarkan pengalaman penulis. Bakteri yang akan digunakan dalam artikel ini adalah Escherichia coli str. K-12 (Catatan : Penting untuk mengetahui strain mikroba yang akan digunakan, karena pada dasarnya ada beberapa perbedaan pada setiap strain dalam metabolismenya).
- Langkah 1 : Siapkan data yang diperlukan untuk pemrosesan menggunakan aplikasi bioinformatika RAST. Data dapat diperoleh dari NCBI. Usahakan data berbentuk whole genome/ complete genome seperti pada Gambar Langkah 1. Download data dalam bentuk Genbank (GB) atau FASTA dengan cara klik Send to à centang File à Lalu pilih Genbank/FASTA.
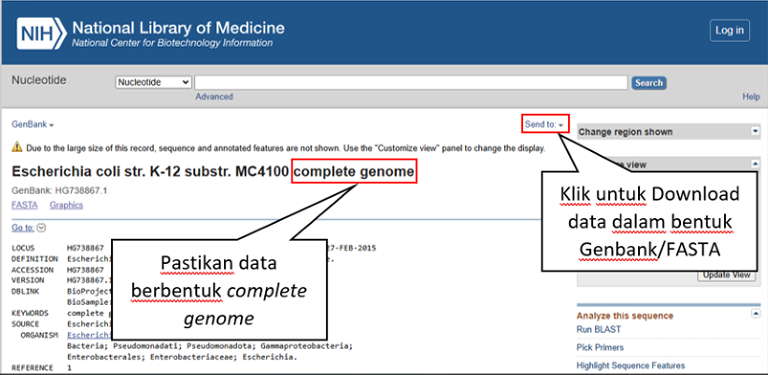
Langkah 1. Pencarian Data pada NCBI, Sumber : https://www.ncbi.nlm.nih.gov/ - Lakukan registrasi terlebih dahulu pada situs RAST . Selanjutnya akan ada notifikasi melalui email yang telah terdaftar terkait username dan password untuk login ke situs RAST. Setelah berhasil login, tampilan utama RAST akan terlihat seperti gambar pada Langkah 3

- Scroll ke bawah, anda akan menemukan kolom file upload. Upload data anda (dalam ekstensi .gb atau fasta) lalu klik “use this data and go to step 2”

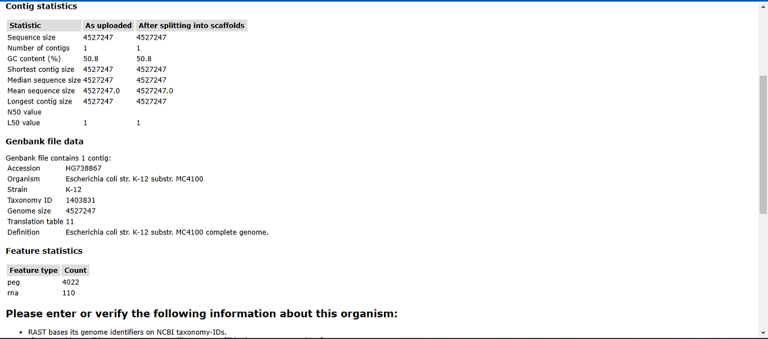
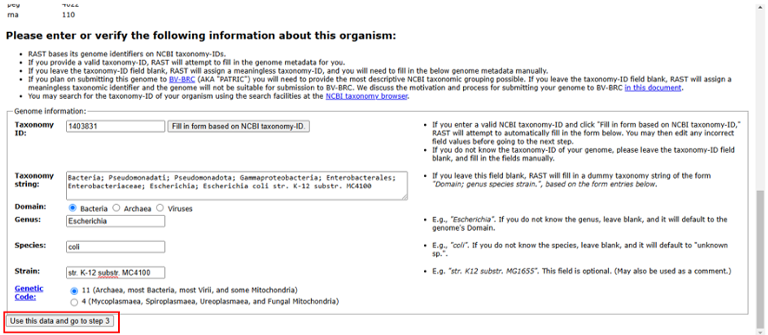
- Cek Kembali informasi data yang telah dimasukkan, pastikan informasi data sesuai dengan organisme yang ingin diteliti. Setelah data dirasa sesuai, lanjutkan ke tahap selanjutnya dengan mengklik “use this data and go to step 3”
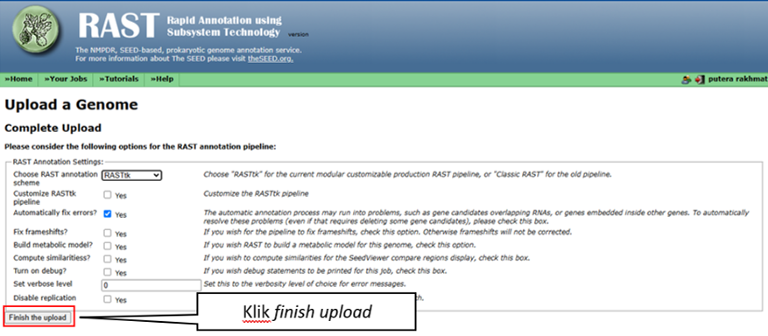
- Biarkan pengaturan seperti pada default (Gambar Langkah 6) yang tersedia, lalu klik finish upload
Tunggu hingga proses selesai seperti pada gambar Langkah 7
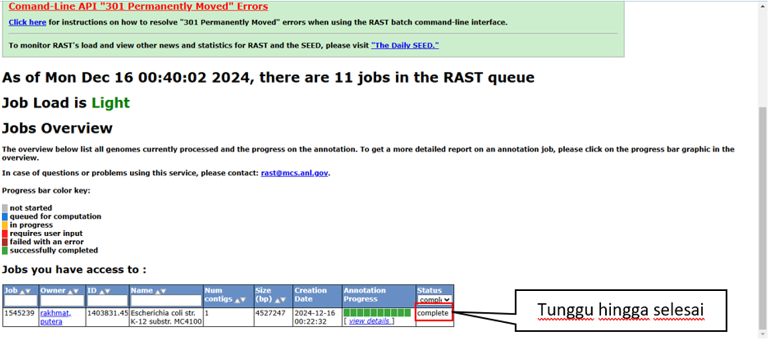
- Hasil pemrosesan akan terlihat seperti gambar Langkah 8 dan Langkah 9, tekan browse annotated genome in SEED Viewer untuk melihat genom yang telah teranotasi
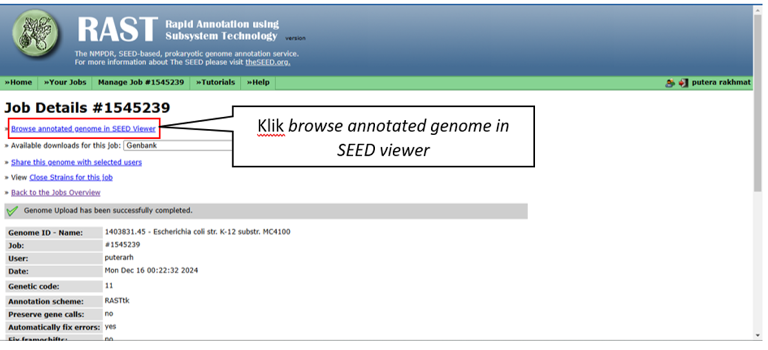
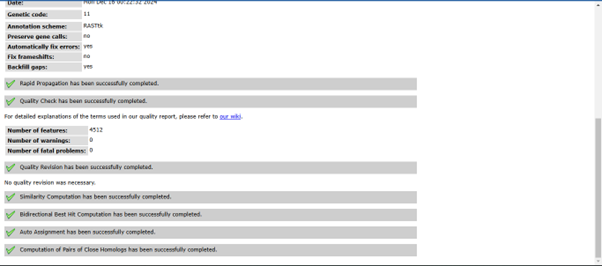
Gambar berikut adalah hasil anotasi genom pada SEED Viewer
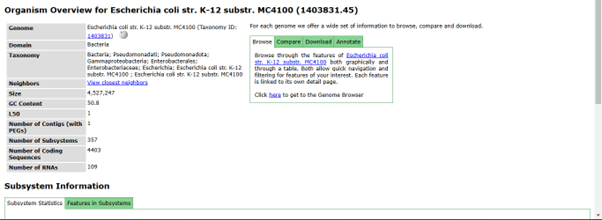
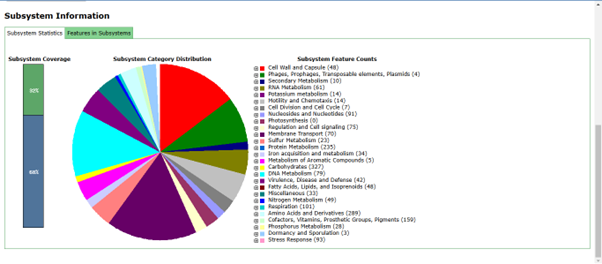
Hasil anotasi menunjukkan beberapa bagian diantaranya :
- Deskripsi umum organisme yang dimasukkan
- Subsystem Statistic : menunjukkan sebesar 32 % dari gene berhasil dianotasikan dan terlihat memiliki fungsi spesifik, sedangkan 68 % menunjukkan gene yang tidak dianotasikan, yang diperkirakan belum diketahui fungsi atau belum dikarakterisasi.
- Pie chart menunjukkan distribusi relative dari gen yang dianotasi berdasarkan fungsinya. Beberapa yang terlihat seperti :
- kelompok karbohidrat (327 gen) yang menunjukkan kemampuan metabolisme karbohidrat yang kuat pada organisme, seperti produksi energi dan fungsi struktural
- metabolisme protein (235 gen)
- biosintesis asam amino dan turunannya (92 gen)
- metabolisme DNA (61 gen)
Informasi lebih lengkap dari gen yang dianotasi tersedia pada tab “feature in subsystem”
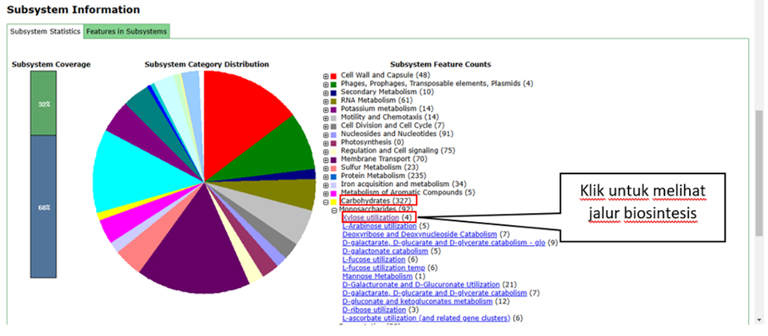
Jika ingin melihat jalur biosintesis dari masing masing gen yang dianotasikan, bisa mengklik salah satu tab yang tersedia (contoh : carbohydrate) lalu klik gen yang ingin dilihat jalur biosintesisnya (contoh : xylose)
Berikut tampilan jalur biosintesis Xylosa setelah diklik. Dimana :
- Diagram : memvisualisasikan jalur metabolisme dan komponennya.
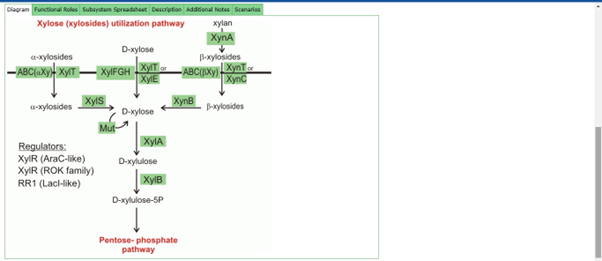
- Functional Roles : untuk memahami fungsi spesifik gen yang terlibat.
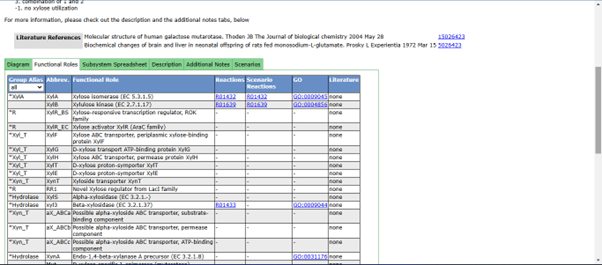
- Subsystem Spreadsheet : untuk verifikasi keberadaan gen dan lokasi genomnya.
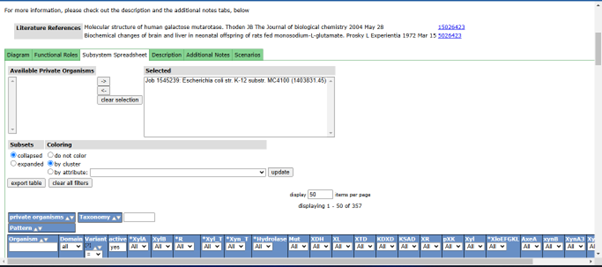
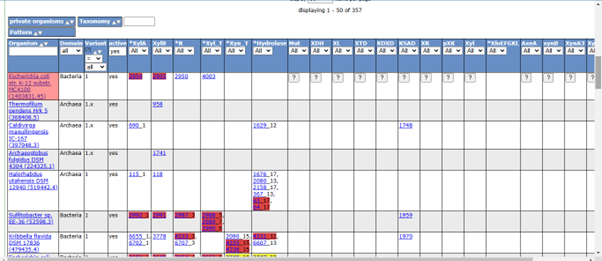
- Read Description : untuk mempelajari pentingnya jalur ini secara biologis
- Additional Notes and Scenario : untuk mendalami pemahaman tentang prediksi dan anotasi eksperimental.
Bagaimana Saya Menggunakan RAST sebagai Seorang Engineer?
sebagai seorang Engineer, RAST akan sangat bermanfaat terutama untuk :
- memahami fungsi gen dan jalur metabolisme dalam organisme yang digunakan (misalnya E. coli, Pseudomonas, Bacillus).
- merancang pabrik sel mikroba yang menghasilkan biofuel, enzim, bioplastik, dll.
dan fungsi lain yang terkait.
Bagaimana? setelah membaca artikel tersebut, apakah anda tertarik untuk mencoba aplikasi ini?
Sumber :
-
Alberts, B., Johnson, A., Lewis, J., Raff, M., Roberts, K., & Walter, P. (2014). Molecular biology of the cell (6th ed.). Garland Science.
-
Aziz, R. K., Bartels, D., Best, A. A., DeJongh, M., Disz, T., Edwards, R. A., ... & Zagnitko, O. (2008). The RAST Server: Rapid annotations using subsystems technology. BMC Genomics, 9(1), 75.
-
Brettin, T., Davis, J. J., Disz, T., Edwards, R. A., Gerdes, S., Olsen, G. J., ... & Xia, F. (2015). RASTtk: A modular and extensible implementation of the RAST technology. Scientific Reports, 5, 8365.
-
Overbeek, R., Olson, R., Pusch, G. D., Olsen, G. J., Davis, J. J., Disz, T., ... & Xia, F. (2014). The SEED and the Rapid Annotation of microbial genomes using Subsystems Technology (RAST). Nucleic Acids Research, 42(D1), D206–D214.
-
Steinbiss, S., & Kurtz, S. (2012). AnnotationSketch: A genome annotation drawing library. Bioinformatics, 28(12), 1533–1534.
0 Komentar
Artikel Terkait



